Anteils- und Boniturdaten auswerten
In dieser Woche geht es gleich um zwei Datentypen: Anteils- und Bonitur-Daten. Anteilsdaten können mit generalisierten linearen Modellen ausgewertet werden, die Sie beim letzten Mal schon kennengelernt haben - allerdings mit zwei kleinen Änderungen. Bonitur-Daten sind im Gartenbau ziemlich häufig, sind allerdings in Bezug auf die Auswertung ein eher undankbarer Datentyp: oft ist es schwierig bis unmöglich, sie durch Verteilungen wie der Normalverteilung zu charakterisieren. Deshalb wiedersetzen sie sich auch den Auswertungen, die auf solchen Verteilungen beruhen.
Am Ende dieses Kapitels wissen Sie
- wie die abhängige Variable in einem GLM zu Anteilsdaten generiert wird
- mit welchem Modell und welchem Test Sie Anteilsdaten auswerten, um signifikante Unterschiede zwischen den Behandlungen zu identifizieren
- wie man die etwas störrischen Bonitur-Daten zähmen, plotten und auswerten kann.
Anteilsdaten auswerten
Im Keimungsversuch mit Radis und Bohnen auf zwei verschiedenen Substraten haben Sie Anteilsdaten aufgenommen: wie viele der 80 ausgesäten Samen sind gekeimt? Die csv-Dateien finden Sie hier: https://ecampus.uni-bonn.de/goto_ecampus_file_2037606_download.html
Wie Sie sich schon denken können, erfüllt auch dieser Datentyp nicht die Annahmen, die für eine ‘normale’ Anova gegeben sein müssen. Zum Glück können wir wieder auf die generalisierten linearen Modelle zurückgreifen, allerdings mit zwei kleinen Änderungen. Aber fangen wir von vorne an.
Zuerst laden wir das Paket ‘tidyverse’, das uns zum Beispiel das Umstrukturieren von Datensätzen erleichtert, lesen die Daten ein (bei mir sind sie wieder im Ordner ‘data’ innerhalb des Ordners, in dem sich das r-Skript befindet gespeichert) und kontrollieren, ob alles geklappt hat:
library(tidyverse)
## ── Attaching packages ─────────────────────────────────────── tidyverse 1.3.0 ──
## ✓ ggplot2 3.2.1 ✓ purrr 0.3.4
## ✓ tibble 3.0.3 ✓ dplyr 1.0.0
## ✓ tidyr 1.0.0 ✓ stringr 1.4.0
## ✓ readr 1.3.1 ✓ forcats 0.4.0
## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()
Ansaaten <- read.csv("data/Ansaaten.csv", header = T)
head(Ansaaten)
str(Ansaaten)
summary(Ansaaten)
Um uns die Keimungsraten genauer anzuschauen, erstellen wir einen neuen Datensatz ‘Keimung’, indem wir mit dem pipe-Operator %>% aus dem Datensatz ‘Ansaaten’ die entsprechenden Spalten auswählen
Keimung <- Ansaaten %>% select(Art, ID, Substrat, Ausfaelle, Angelaufen)
head(Keimung)
## Art ID Substrat Ausfaelle Angelaufen
## 1 Bohne 1 Presstopf 5 79
## 2 Bohne 2 Presstopf 3 81
## 3 Bohne 3 Presstopf 6 78
## 4 Bohne 4 Presstopf 6 78
## 5 Bohne 5 Presstopf 21 63
## 6 Bohne 6 Presstopf 23 61
Um einen Säulendiagramm zu erstellen, in dem die Anzahl der angelaufenen Samen und der Ausfälle gestapelt dargestellt werden, müssen wir den Datensatz umstrukturieren, um für jede Anzahl ‘Ausfaelle’ oder ‘Angelaufen’ eine eigene Spalte zu bekommen. Dazu nutzen wir die Funktion ‘reshape’. Wichtig sind hierbei die Parameter
- idvar = die Kombination an Plot ID - Behandlungslevels, die eine Zeile in der neuen Datenstruktur definiert
- timevar = Bezeichnung für neue Spalte
- times = das, was in diese Spalte eingetragen werden soll
Keimung_w <- reshape(Keimung,
direction = "long",
varying = list(names(Keimung)[4:5]),
v.names = "Anzahl",
idvar = c("Art", "ID", "Substrat"),
timevar = "Beobachtung",
times = c("Ausfaelle", "Angelaufen"))
head(Keimung_w)
## Art ID Substrat Beobachtung Anzahl
## Bohne.1.Presstopf.Ausfaelle Bohne 1 Presstopf Ausfaelle 5
## Bohne.2.Presstopf.Ausfaelle Bohne 2 Presstopf Ausfaelle 3
## Bohne.3.Presstopf.Ausfaelle Bohne 3 Presstopf Ausfaelle 6
## Bohne.4.Presstopf.Ausfaelle Bohne 4 Presstopf Ausfaelle 6
## Bohne.5.Presstopf.Ausfaelle Bohne 5 Presstopf Ausfaelle 21
## Bohne.6.Presstopf.Ausfaelle Bohne 6 Presstopf Ausfaelle 23
Jetzt können wir den Plot erstellen
ggplot(Keimung_w, aes(x=factor(ID), y=Anzahl, fill=Beobachtung)) +
facet_grid(. ~ Art * Substrat) +
geom_bar(position="stack", stat="identity") +
scale_fill_manual("legend", values = c("Angelaufen" = "darkgreen", "Ausfaelle" = "lightgrey")) +
geom_col(position = position_stack(reverse = TRUE))
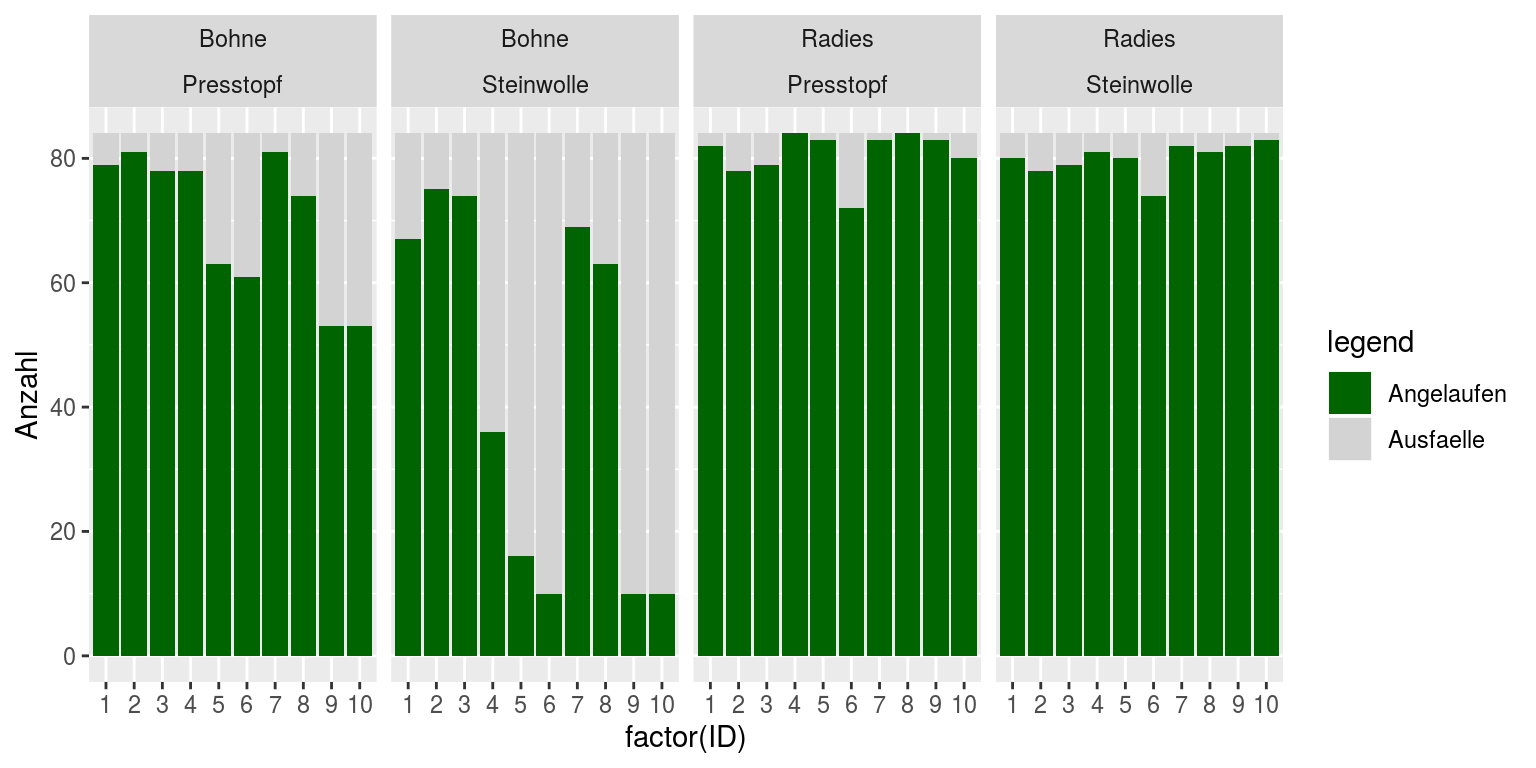
Wir sehen, dass die Erfolgsrate bei den Radis unabhängig vom Substrat sehr hoch ist. Bei den Bohnen gibt es mehr Ausfälle, insbesondere bei denen auf Steinwolle ausgesäten.
Sind diese Unterschiede signifikant? Um das zu testen, können wir wieder die generalisierten linearen Modelle fitten und mittels der anova-Funktion vergleichen. Bei Anteilsdaten wie diesen erfolgt davor allerdings noch ein Vorbereitungsschritt: Wir generieren aus den beiden Spalten ‘Angelaufen’ und ‘Ausfaelle’ eine einzige abhängige Variable, die wir ‘Keimungsrate’ nennen. Es ist wichtig, die Rate nicht selber auszurechnen: ‘1 von 10 Samen gekeimt’ hat im Modell weniger Gewicht als ‘10 von 100’ Samen gekeimt. Stattdessen verwenden wir die Funktion ‘cbind()’ :
Ansaaten$Keimungsrate <- cbind(Ansaaten$Angelaufen, Ansaaten$Ausfaelle)
Dann können wir die Funktion glm() wie bekannt nutzen. Bei Anteilsdaten nutzen wir als family ‘binomial’
Modell1 <- glm(Keimungsrate ~ Substrat * Art, family = binomial, data = Ansaaten)
summary(Modell1)
##
## Call:
## glm(formula = Keimungsrate ~ Substrat * Art, family = binomial,
## data = Ansaaten)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -7.6302 -1.6393 0.6342 2.5570 7.4933
##
## Coefficients:
## Estimate Std. Error z value Pr(>|z|)
## (Intercept) 1.61803 0.09285 17.427 < 2e-16 ***
## SubstratSteinwolle -1.57041 0.11570 -13.574 < 2e-16 ***
## ArtRadies 1.61079 0.20275 7.945 1.95e-15 ***
## SubstratSteinwolle:ArtRadies 1.33731 0.26856 4.980 6.37e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for binomial family taken to be 1)
##
## Null deviance: 1266.76 on 39 degrees of freedom
## Residual deviance: 561.64 on 36 degrees of freedom
## AIC: 705.33
##
## Number of Fisher Scoring iterations: 5
Die summary des Modells zeigt, dass die Daten overdispersed sind. Auch hier gibt es für diesen Fall die Möglichkeit als family ‘quasibinomial’ anzugeben:
Modell1 <- glm(Keimungsrate ~ Substrat * Art, family = quasibinomial, data = Ansaaten)
Zunächst lassen wir die Interaktion zwischen Substrat und Art weg, indem wir das Multiplikationszeichen durch ein Plus ersetzen
Modell2 <- glm(Keimungsrate ~ Substrat + Art, family = quasibinomial, data = Ansaaten)
Jetzt vergleichen wir die Modelle mittels anaova(). Als Test muss jetzt der F-Test angegeben werden
anova(Modell1, Modell2, test = "F")
## Analysis of Deviance Table
##
## Model 1: Keimungsrate ~ Substrat * Art
## Model 2: Keimungsrate ~ Substrat + Art
## Resid. Df Resid. Dev Df Deviance F Pr(>F)
## 1 36 561.64
## 2 37 584.97 -1 -23.326 1.603 0.2136
Wie wir sehen, ist die Interaktion nicht signifikant (p > 0.05) Also vereinfachen wir das Modell weiter und nehmen jetzt das Substrat als erklärende Variable raus
Modell3 <- glm(Keimungsrate ~ Art, family = quasibinomial, data = Ansaaten)
# Wieder vergleichen:
anova(Modell2, Modell3, test = "F")
## Analysis of Deviance Table
##
## Model 1: Keimungsrate ~ Substrat + Art
## Model 2: Keimungsrate ~ Art
## Resid. Df Resid. Dev Df Deviance F Pr(>F)
## 1 37 584.97
## 2 38 767.96 -1 -182.99 11.644 0.001573 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Das Substrat hat einen hochsignifikanten Effekt auf die Keimungsrate. Als letztes testen wir noch die Art
Modell4 <- glm(Keimungsrate ~ Substrat, family = quasibinomial, data = Ansaaten)
Jetzt müssen wir Modell4 natürlich gegen Modell2 testen. Die Modelle müssen immer ‘genested’ sein, sonst kann kein Signifikanzniveau berechnet werden
anova(Modell2, Modell4, test = "F")
## Analysis of Deviance Table
##
## Model 1: Keimungsrate ~ Substrat + Art
## Model 2: Keimungsrate ~ Substrat
## Resid. Df Resid. Dev Df Deviance F Pr(>F)
## 1 37 584.97
## 2 38 1108.34 -1 -523.38 33.303 1.283e-06 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Auch die Art hat einen hochsignifikanten Einfluss auf die Keimungsrate. Damit ist die Analyse der Keimungsraten abgeschlossen.
Bonitur-Daten
Bonitur-Daten sind recht speziell, weil es keine metrischen Daten sind, sondern Ordinal-Daten und darüber hinaus auch einigermaßen subjektiv. Trotzdem sind hierfür Methoden zur Auswertung entwickelt worden. Sie stammen aus der Psychologie, in der häufig Untersuchungen stattfinden, bei denen Personen zum Beispiel ihre Zufriedenheit auf einer Skala von 1 bis 5 bewerten sollen.
Wir schauen uns dazu exemplarisch die Bonitur-Daten zur Entwicklung der Bohnen an. Dazu filtern wir den Datensatz Ansaaten, indem wir nur die Zeilen nehmen, in denen das Argument Art == “Bohne” zutrifft (das doppelte Gleichzeichen wird immer benutzt, wenn eine Abfrage gemacht wird, ein einfaches Gleichzeichen ist in R eine Zuweisung also synonym mit dem Pfeil <- ). Mit dem Pipe-Operator %>% verbinden wir diese Auswahl mit der Auswahl der Spalten, die wir mit der Funktion select() machen. Hier können wieder einfach die header (=Spaltenüberschriften) der benötigten Spalten angegeben werden. Vorher laden wir noch zwei Pakete, die für die Auswertung von Ordinal-Daten entwickelt wurden
library(likert)
## Loading required package: xtable
##
## Attaching package: 'likert'
## The following object is masked from 'package:dplyr':
##
## recode
library(ordinal)
##
## Attaching package: 'ordinal'
## The following object is masked from 'package:dplyr':
##
## slice
Bohne_Entw_l <- filter(Ansaaten, Art == "Bohne") %>% select(Substrat, ID, Entwicklung)
head(Bohne_Entw_l)
## Substrat ID Entwicklung
## 1 Presstopf 1 8
## 2 Presstopf 2 5
## 3 Presstopf 3 8
## 4 Presstopf 4 8
## 5 Presstopf 5 4
## 6 Presstopf 6 5
Als erstes sollten die Daten wieder geplottet werden. Bei Bonitur-Daten eignen sich Histogramme ab besten. Um das zu tun, müssen die Bonitur-Noten in Faktoren umgewandelt werden. Für die weitere Analyse geben wir auch direkt an, wieviele Levels es gibt und dass sie eine aussagekräftige Reihenfolge haben (ordered = TRUE):
Bohne_Entw_l$Entwicklung <- factor(Bohne_Entw_l$Entwicklung, ordered = TRUE, levels = 1:9)
Jetzt können die Histogramme erstellt werden:
ggplot(Bohne_Entw_l, aes(Entwicklung)) +
geom_bar() +
facet_wrap(~Substrat, ncol=1) +
xlab("Bonitur Note") +
ylab("Anzahl")
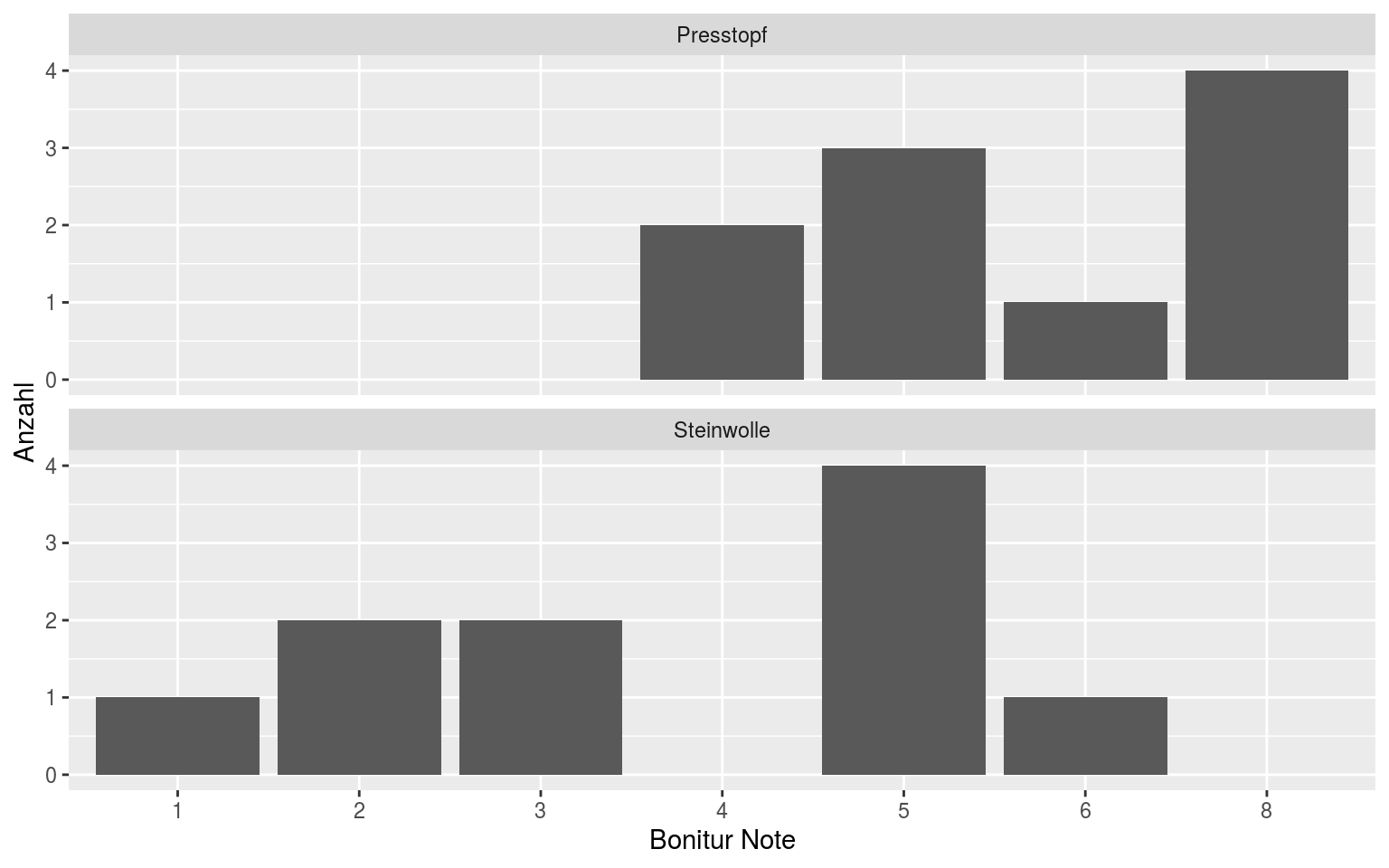
Das package ‘likert’ stellt einige Plot-Funktionen zur Verfügung, die speziell für Bonitur- und andere Typen von likert-Daten geeignet sind. Dazu brauchen wir die Daten allerdings im ‘wide’ Format. Im Moment haben wir die Daten im ‘long’ Format: es gibt eine eigene Spalte für das Substrat, sodass ‘Presstopf’ und ‘Steinwolle’ je 10 Mal genannt werden. Wir nutzen wieder die Funktion ‘reshape’ wie oben, jetzt allerdings in umgekehrter Richtung, vom ‘long’ zum ‘wide’ Format:
Bohne_Entw_w <- reshape(Bohne_Entw_l, v.names="Entwicklung", idvar = "ID", timevar = "Substrat", direction="wide")
head(Bohne_Entw_w)
## ID Entwicklung.Presstopf Entwicklung.Steinwolle
## 1 1 8 5
## 2 2 5 5
## 3 3 8 5
## 4 4 8 3
## 5 5 4 2
## 6 6 5 1
‘idvar’ sind jetzt also die Zeilenbezeichnungen, ‘timevar’ die Spaltenbezeichnungen, ‘v.names’ die eigentlichen Werte.
Als nächstes müssen wir ein sogenanntes ‘likert-Objekt’ aus den beiden Spalten mit den Bonitur-Noten erstellen, dann kann die plot-Funktion des likert-packages genutzt werden:
Bohne_Entw_likert = likert(Bohne_Entw_w[2:3])
plot(Bohne_Entw_likert)
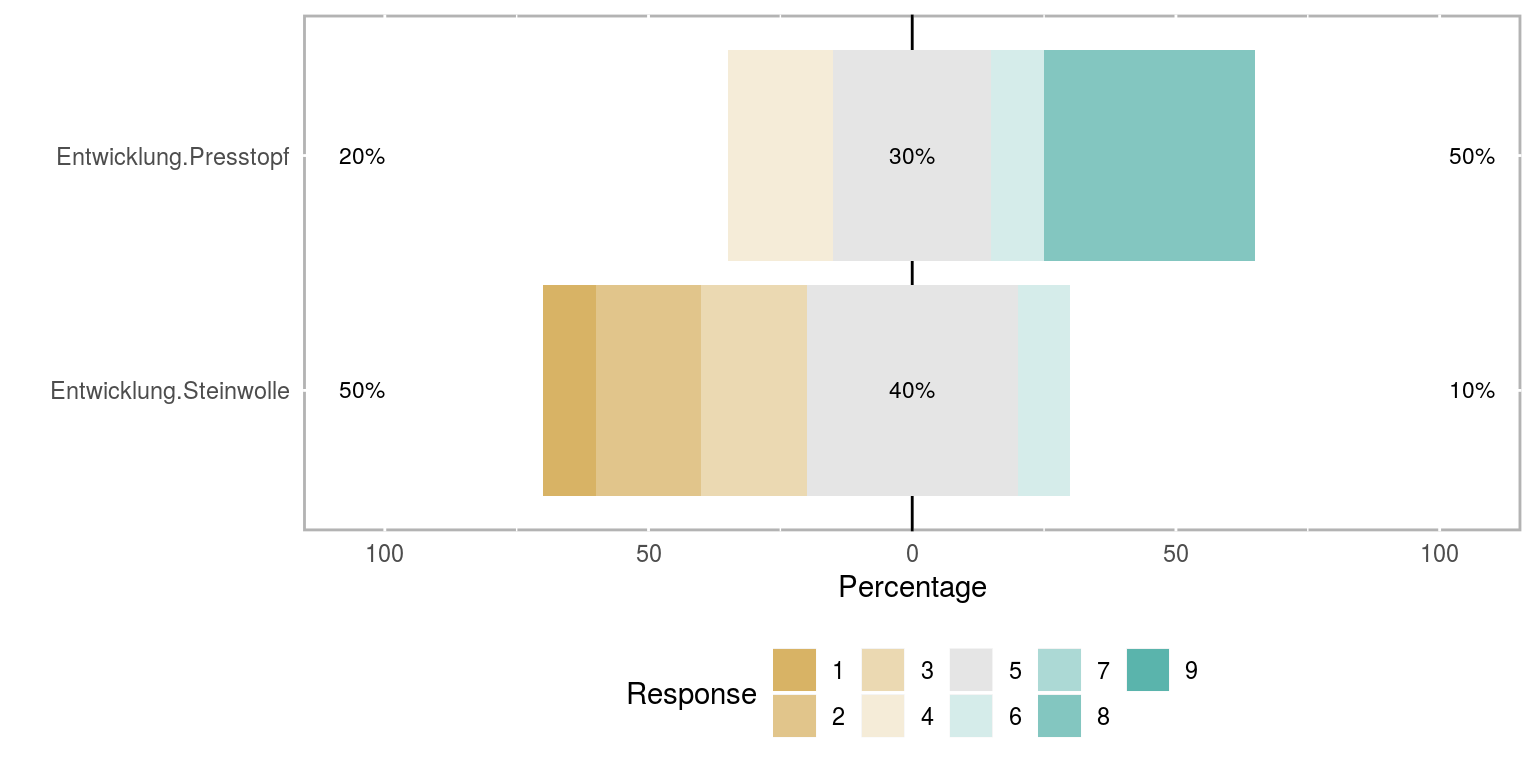
Dies ist eine andere - etwas kompaktere - Darstellung der gleichen Daten. Die %-Werte geben an, wieviel % im schlechten Bereich (1-3), im mittleren Bereich (4-6) und im guten Bereich (7 - 9) waren.
Bonitur-Daten sind Ordinal-Daten und haben damit andere Eigenschaften als metrische Daten. Wie oben erwähnt erfüllen sie häufig nicht die Annahmen, die für eine normale Varianzanalyse zutreffen müssen. Wir greifen hier auf ‘cumulative link models’ (CLM) aus der library ‘ordered’ zurück. CLMs sind im Prinzip das gleiche wie proportional odds model, falls Ihnen dieser Begriff mal über den Weg läuft. Ähnlich wie in den vorherigen beiden Kapiteln formulieren wir wieder ein komplexeres Modell mit Substrat als erklärender Variable und das Null-Modell, indem angenommen wird, dass alle Bohnen sich ungeachtet der Behandlung gleich Entwickeln:
Model1 <- clm(Entwicklung ~ Substrat, data = Bohne_Entw_l, threshold = "equidistant")
Model2 <- clm(Entwicklung ~ 1, data = Bohne_Entw_l, threshold = "equidistant")
Als threshhold geben wir ‘equidistant’ an, weil wir davon ausgehen, dass zum Beispiel der Unterschied in der Entwicklung zwischen den Bonitur-Noten 2 und 3 genauso groß ist, wie der Unterschied zwischen den Noten 3 und 4 usw.
Auch für CLMs gibt es die Funktion anova(). Hier wird allerdings kein t- oder F-Test durchgeführt, sondern ein Chi-square Test, der uns einen p-Wert liefert:
anova(Model1, Model2)
## Likelihood ratio tests of cumulative link models:
##
## formula: link: threshold:
## Model2 Entwicklung ~ 1 logit equidistant
## Model1 Entwicklung ~ Substrat logit equidistant
##
## no.par AIC logLik LR.stat df Pr(>Chisq)
## Model2 2 78.001 -37.000
## Model1 3 73.330 -33.665 6.6711 1 0.009799 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Wie sie sehen, ist der p-Wert etwa 0.01, das heißt, das Substrat hat einen signifikanten Effekt auf die Entwicklung der Bohnen. Hiermit ist auch eine grundlegende Analyse der Bonitur-Daten abgeschlossen.
Ein interessanter Link zum Thema, inklusive Auswertungsmöglichkeiten mit R: ‘Do not use averages with Likert scale data’